sklearn.cluster.AgglomerativeClustering¶
class sklearn.cluster.AgglomerativeClustering(n_clusters=2, *, affinity='euclidean', memory=None, connectivity=None, compute_full_tree='auto', linkage='ward', distance_threshold=None)
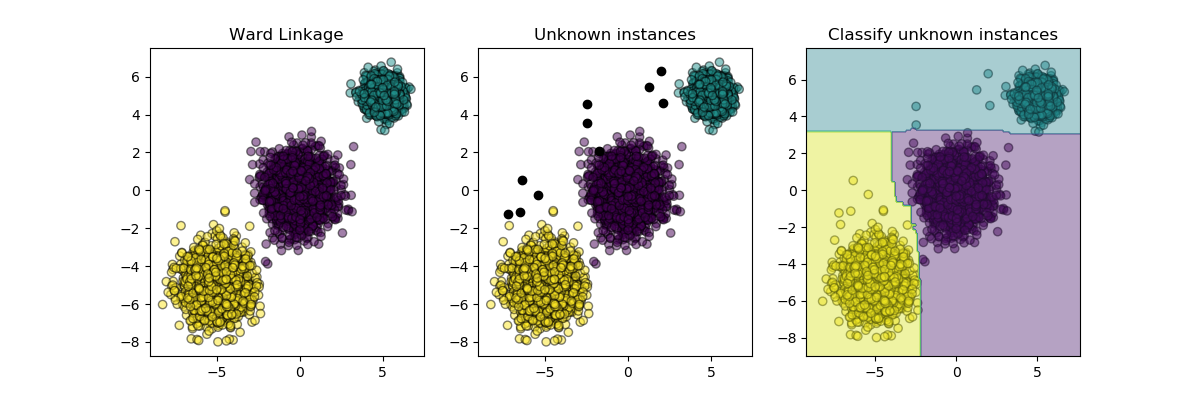
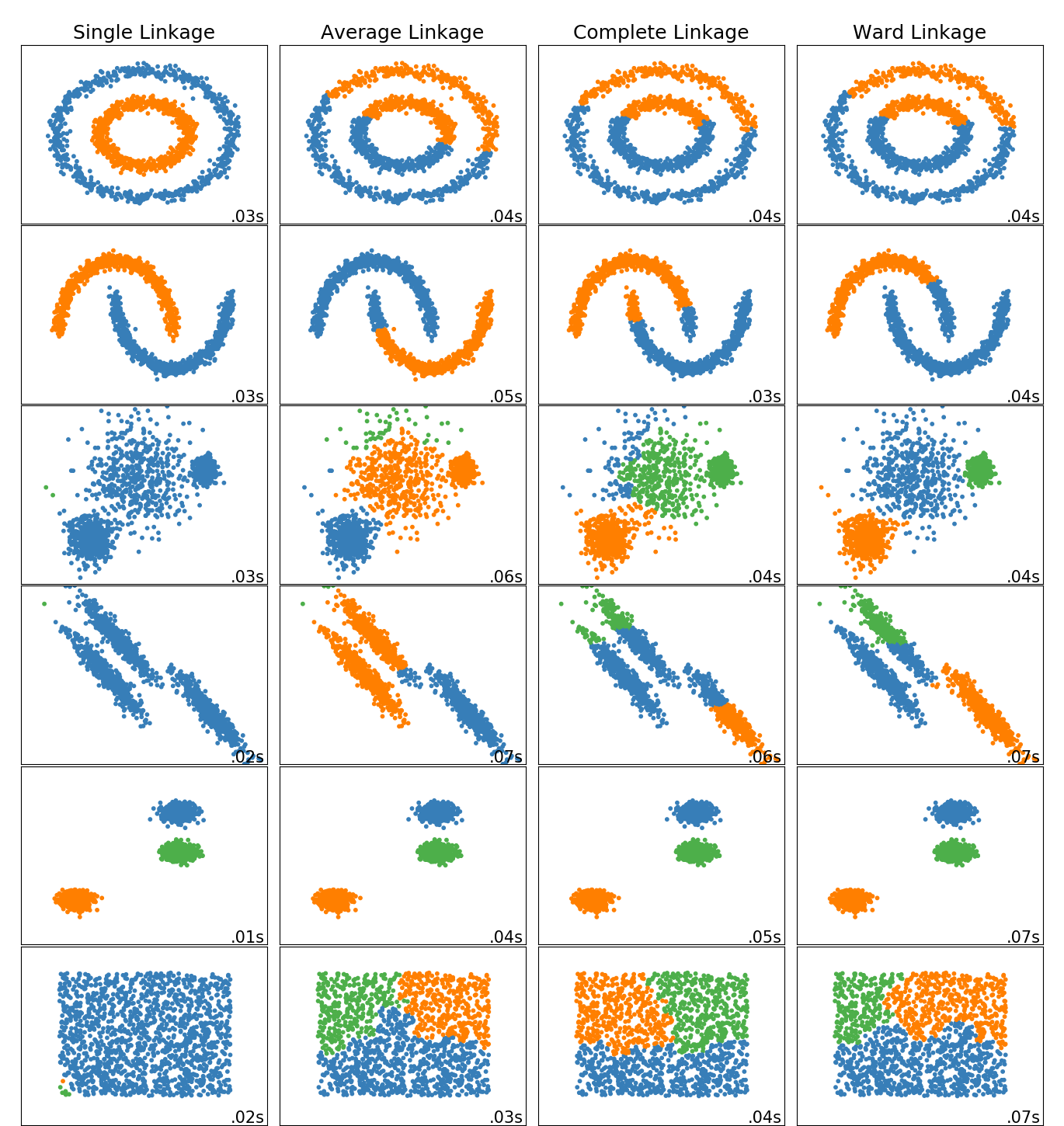
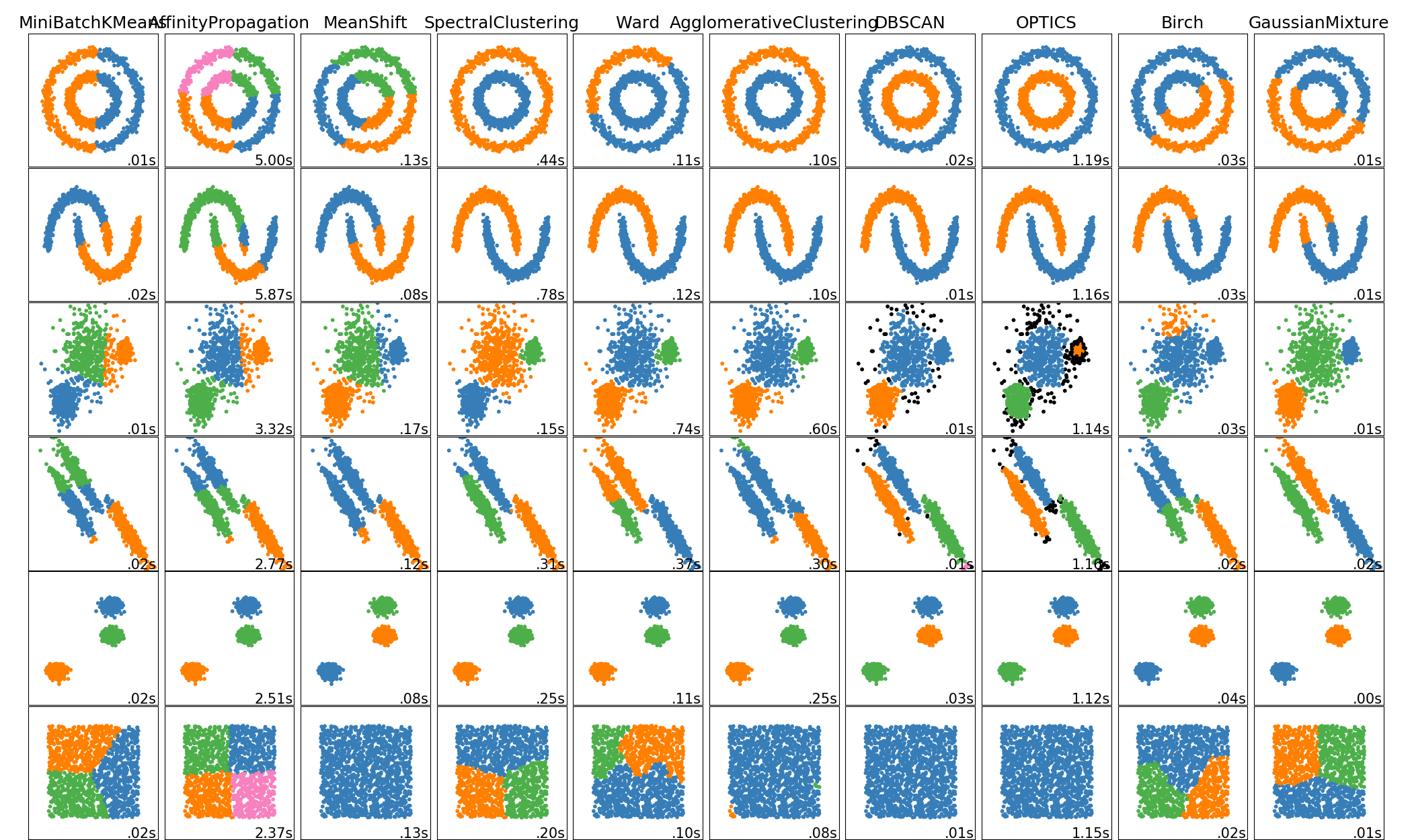
凝聚聚类
递归地合并成对聚类,以最小的方式增加给定的链接距离。
在用户指南中阅读更多内容。
| 参数 | 说明 |
|---|---|
| n_clusters | int or None, default=2 要查找的聚类数目。如果 distance_threshold 不是None, 它就必须是None。 |
| affinity | str or callable, default=’euclidean’ 用于计算连接的度量。可以是“euclidean”, “l1”, “l2”, “manhattan”, “cosine”, 或者“precomputed”。如果linkage是“ward”, 只有“euclidean”是被接受的。 如果“precomputed”,则需要一个距离矩阵(而不是相似矩阵)作为拟合方法的输入。 |
| memory | str or object with the joblib.Memory interface, default=None 用于储存树计算的输出。默认情况下,不执行缓存。如果给出一个字符串,它就是缓存目录的路径。 |
| connectivity | array-like or callable, default=None 连接矩阵。为每个样本定义遵循给定数据结构的相邻样本。这可以是连接矩阵本身,也可以是可调用的,能将数据转换为连接矩阵,例如从kneighbors_graph派生的连接矩阵。默认为None,分层聚类算法是一种非结构化的聚类算法。 |
| compute_full_tree | ‘auto’ or bool, default=’auto’ 提前停止n_clusters树的构建。如果聚类的数量与样本数相比并不少,这对于减少计算时间是非常有用的。此选项仅在指定连接矩阵时才有用。还请注意,当改变聚类数量并使用缓存时,计算所有树可能更有利。如果 distance_threshold是None, 它必须是True。通过默认的compute_full_tree设置是“auto”, 当distance_threshold不是None的时候就等价于True,或者n_clusters的最大值在100~0.02 * n_samples之间。否则,“auto”等同False。 |
| linkage | {“ward”, “complete”, “average”, “single”}, default=”ward” 使用哪种联动标准。该算法将合并聚类,以最小化这一标准。 - Ward最小化会合并的聚类的差异。 - 平均使用两组每次观测的平均距离。 - 完全或最大连接使用两个集合的所有观测值之间的最大距离。 - 单次使用两组所有观测值之间的最小距离。 |
| distance_threshold | float, default=None 连接距离阈值高于该阈值,聚类将不会合并。如果不是None,则 n_clusters必须为None,并且compute_full_tree必须为True。 |
新版本0.21。
| 属性 | 说明 |
|---|---|
| n_clusters_ | int 通过算法找到的聚类数。如果 distance_threshold=None=None,则它将等价于给定的n_clusters。 |
| labels_ | ndarray of shape (n_samples) 每一点的聚类标签 |
| n_leaves_ | int 层次树中的叶子节点数 |
| n_connected_components_ | int 图中连通分量的估计数 新版本0.21中: n_connected_components_被添加以代替n_components_ |
| children_ | array-like of shape (n_samples-1, 2) 每个非叶节点的子节点。小于 n_samples的值对应于原始样本树的叶子。大于或等于n_samples的节点i是一个非叶节点,具有子节点children_[i - n_samples]。或者,在第i次迭代时,将children[i][0]和children[i][1]合并成节点n_samples + i。 |
示例
>>> from sklearn.cluster import AgglomerativeClustering
>>> import numpy as np
>>> X = np.array([[1, 2], [1, 4], [1, 0],
... [4, 2], [4, 4], [4, 0]])
>>> clustering = AgglomerativeClustering().fit(X)
>>> clustering
AgglomerativeClustering()
>>> clustering.labels_
array([1, 1, 1, 0, 0, 0])
方法
| 方法 | 说明 |
|---|---|
fit(self, X[, y]) |
根据特征或距离矩阵进行层次聚类拟合 |
fit_predict(self, X[, y]) |
根据特征或距离矩阵拟合分层聚类,并返回聚类标签。 |
get_params(self[, deep]) |
获取此估计器的参数 |
set_params(self, **params) |
设置此估计器的参数 |
__init__(self, n_clusters=2, *, affinity='euclidean', memory=None, connectivity=None, compute_full_tree='auto', linkage='ward', distance_threshold=None)
初始化self。请参阅help(type(self))以获得准确的说明。
fit(self, X, y=None)
根据特征或距离矩阵进行层次聚类拟合
| 参数 | 说明 |
|---|---|
| X | array-like, shape (n_samples, n_features) or (n_samples, n_samples) 要聚类的训练实例,或实例之间的距离, 如果 affinity='precomputed' |
| y | Ignored 未使用,在此按约定呈现为API一致性。 |
| 返回值 | 说明 |
|---|---|
| self | - |
fit_predict(self, X, y=None)
根据特征或距离矩阵进行层次聚类拟合
| 参数 | 说明 |
|---|---|
| X | array-like, shape (n_samples, n_features) or (n_samples, n_samples) 要聚类的训练实例,或实例之间的距离, 如果 affinity='precomputed' |
| y | Ignored 未使用,在此按约定呈现为API一致性。 |
| 返回值 | 说明 |
|---|---|
| labels | ndarray, shape (n_samples,) 聚类标签 |
get_params(self, deep=True)
获取此估计器的参数
| 表格 | 说明 |
|---|---|
| deep | bool, default=True 如果为True,则将返回此估计器的参数和所包含的作为估计量的子对象。 |
| 返回值 | 说明 |
|---|---|
| params | mapping of string to any 映射到其值的参数名称 |
set_params(self, **params)
设置此估计器的参数
该方法适用于简单估计器以及嵌套对象(例如pipelines)。后者具有表单的 <component>__<parameter>参数,这样就可以更新嵌套对象的每个组件。
| 参数 | 说明 |
|---|---|
| **params | dict 估计器参数 |
| 返回值 | 说明 |
|---|---|
| self | object 估计器实例 |