分类器校准的比较¶
举例说明分类器预测概率的校准。
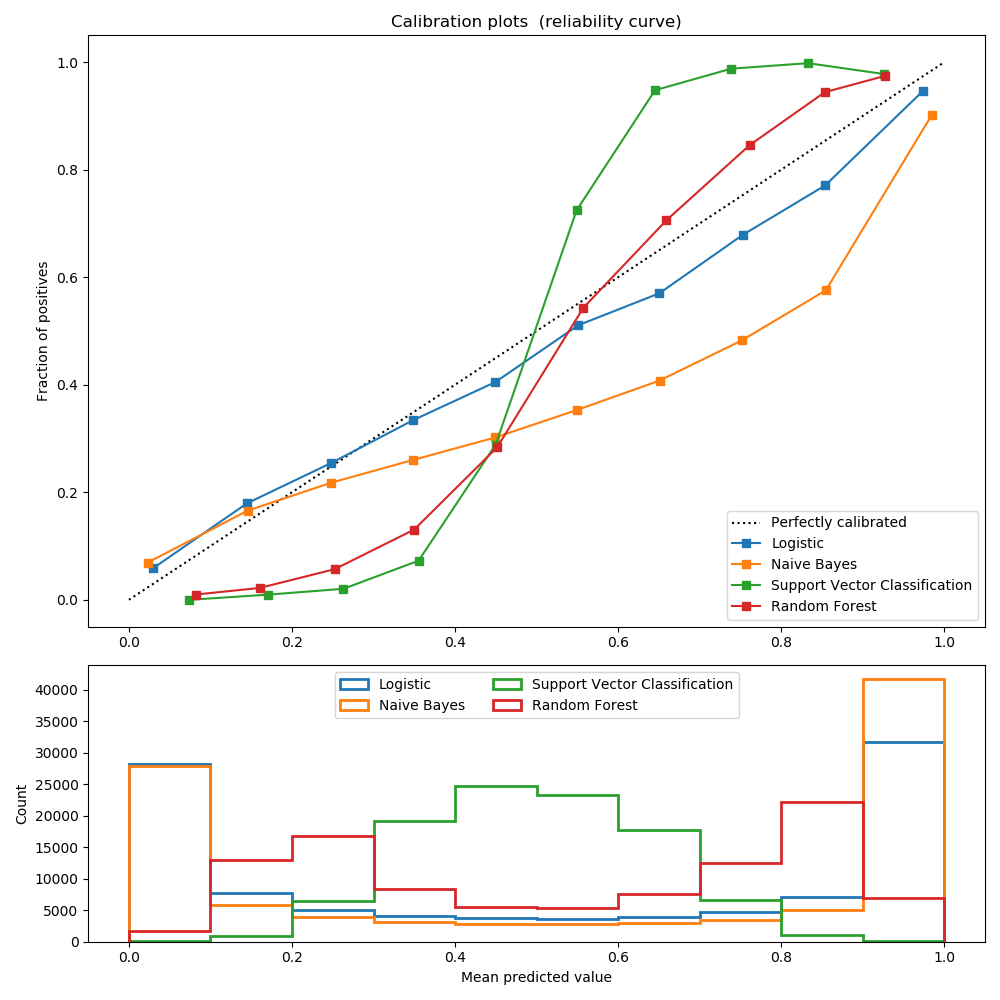
经过良好校准的分类器是概率分类器,它的输出可以直接解释为置信度。例如,经过良好校准的二分类器应该对样本进行分类,这个样本被给予的predict_proba值接近0.8,就意味着大约80%的把握属于正类。
逻辑回归返回良好的校准预测,因为它直接优化对数损失。相反,其他方法返回有偏概率,每种方法有不同的偏差:
GaussianNaiveBayes倾向于将概率推到0或1(注意直方图中的计数)。这主要是因为它假设在给定类的情况下,特征是有条件独立的,而在包含2个冗余特征的数据集中则不是这样。 随机森林分类器显示相反的行为:直方图显示接近峰值。0.2和0.9的概率,而概率接近0或1是非常罕见的。Nculescu-Mizil和Caruana [1]给出了对此的解释:“装袋和随机森林等方法--从一组基本模型的平均预测做出的预测接近0和1是很难的,因为底层基本模型中的方差会导致预测偏差,这些预测值本应该是0或者1, 但是却偏离了这些值。由于预测仅限于区间[0,1],由方差引起的误差往往是近0和1的单边误差。例如,如果一个模型应该对一个情况预测p=0,那么bagging法可以实现的唯一方法就是所有bagging中的树都预测为零。如果将噪声加到bagging中的树木上,这种噪声会导致一些树预测值大于0,从而使bagging集合的平均预测值从0偏离。我们在随机森林中观察到的这种影响最强烈,因为使用随机森林训练的基层树由于特征子集而具有较高的方差。“结果表明,校准曲线呈现出sigmoid形状,表明分类器可以更多地信任它的“直觉”,并返回接近0或1的典型概率。 支持向量分类(SVC)显示了一个比随机森林分类器更加sigmoid形状的曲线,这是典型的最大化间隔方法(比较Nculescu-Mizil和Caruana [1]),重点是关注那些接近决策边界的硬样本(支持向量)。
参考
1(1,2) Predicting Good Probabilities with Supervised Learning, A. Niculescu-Mizil & R. Caruana, ICML 2005
print(__doc__)
# Author: Jan Hendrik Metzen <jhm@informatik.uni-bremen.de>
# License: BSD Style.
import numpy as np
np.random.seed(0)
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.naive_bayes import GaussianNB
from sklearn.linear_model import LogisticRegression
from sklearn.ensemble import RandomForestClassifier
from sklearn.svm import LinearSVC
from sklearn.calibration import calibration_curve
X, y = datasets.make_classification(n_samples=100000, n_features=20,
n_informative=2, n_redundant=2)
train_samples = 100 # Samples used for training the models
X_train = X[:train_samples]
X_test = X[train_samples:]
y_train = y[:train_samples]
y_test = y[train_samples:]
# Create classifiers
lr = LogisticRegression()
gnb = GaussianNB()
svc = LinearSVC(C=1.0)
rfc = RandomForestClassifier()
# #############################################################################
# Plot calibration plots
plt.figure(figsize=(10, 10))
ax1 = plt.subplot2grid((3, 1), (0, 0), rowspan=2)
ax2 = plt.subplot2grid((3, 1), (2, 0))
ax1.plot([0, 1], [0, 1], "k:", label="Perfectly calibrated")
for clf, name in [(lr, 'Logistic'),
(gnb, 'Naive Bayes'),
(svc, 'Support Vector Classification'),
(rfc, 'Random Forest')]:
clf.fit(X_train, y_train)
if hasattr(clf, "predict_proba"):
prob_pos = clf.predict_proba(X_test)[:, 1]
else: # use decision function
prob_pos = clf.decision_function(X_test)
prob_pos = \
(prob_pos - prob_pos.min()) / (prob_pos.max() - prob_pos.min())
fraction_of_positives, mean_predicted_value = \
calibration_curve(y_test, prob_pos, n_bins=10)
ax1.plot(mean_predicted_value, fraction_of_positives, "s-",
label="%s" % (name, ))
ax2.hist(prob_pos, range=(0, 1), bins=10, label=name,
histtype="step", lw=2)
ax1.set_ylabel("Fraction of positives")
ax1.set_ylim([-0.05, 1.05])
ax1.legend(loc="lower right")
ax1.set_title('Calibration plots (reliability curve)')
ax2.set_xlabel("Mean predicted value")
ax2.set_ylabel("Count")
ax2.legend(loc="upper center", ncol=2)
plt.tight_layout()
plt.show()
脚本的总运行时间:(0分1.370秒)




