稀疏逆协方差估计¶
使用 GraphicalLasso 估计器从少量样本中学习协方差和稀疏精度。
要估计概率模型(如高斯模型),估计精度矩阵,即逆协方差矩阵,与估计协方差矩阵一样重要。实际上,高斯模型是由精度矩阵参数化的。
为了在良好的恢复条件下,我们从一个稀疏逆协方差矩阵模型中对数据进行了采样。此外,我们还确保数据不存在太大的相关性(限制了精度矩阵的最大系数),并确保在精度矩阵中有一个无法恢复的小系数。此外,通过少量的观测,可以更容易地恢复相关矩阵而不是协方差,因此我们对时间序列进行了缩放。
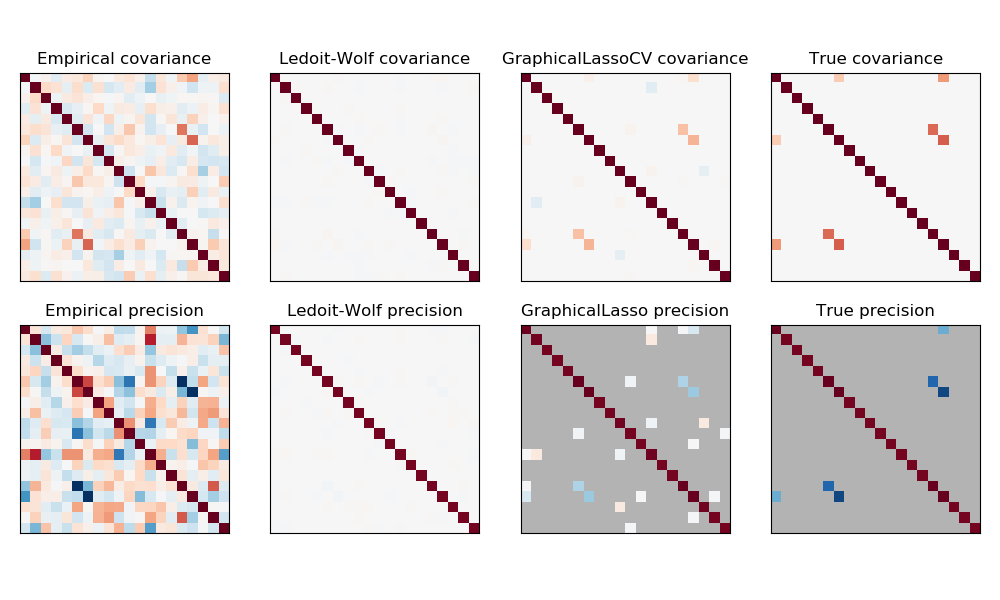
在这里,样本数略大于维数,因此经验协方差仍然是可逆的。然而,由于观测结果有很强的相关性,经验协方差矩阵是不受限制的的,因此它的逆--经验精度矩阵--离实际真是情况很远。
如果我们使用l2 shrinkage,就像 Ledoit-Wolf 估计那样,由于样本数量很小,我们需要进行大量的收缩。因此,Ledoit-Wolf的精度相当接近真实精度,即离对角线不远,但偏离对角线的结构是丢失的。
l1-惩罚的估计器可以恢复这种非对角结构的一部分。它学习了稀疏的精度。它无法恢复精确的稀疏模式:它检测到太多的非零系数。然而,估计的L1的最高非零系数对应于真是情况中的非零系数。最后,L1精度估计的系数偏向于零:由于惩罚,它们都小于相应的真实值,如图中所示。
注意,精确矩阵的颜色范围被调整以提高图形的可读性。没有显示经验精度的全部范围。
print(__doc__)
# author: Gael Varoquaux <gael.varoquaux@inria.fr>
# License: BSD 3 clause
# Copyright: INRIA
import numpy as np
from scipy import linalg
from sklearn.datasets import make_sparse_spd_matrix
from sklearn.covariance import GraphicalLassoCV, ledoit_wolf
import matplotlib.pyplot as plt
# #############################################################################
# Generate the data
n_samples = 60
n_features = 20
prng = np.random.RandomState(1)
prec = make_sparse_spd_matrix(n_features, alpha=.98,
smallest_coef=.4,
largest_coef=.7,
random_state=prng)
cov = linalg.inv(prec)
d = np.sqrt(np.diag(cov))
cov /= d
cov /= d[:, np.newaxis]
prec *= d
prec *= d[:, np.newaxis]
X = prng.multivariate_normal(np.zeros(n_features), cov, size=n_samples)
X -= X.mean(axis=0)
X /= X.std(axis=0)
# #############################################################################
# Estimate the covariance
emp_cov = np.dot(X.T, X) / n_samples
model = GraphicalLassoCV()
model.fit(X)
cov_ = model.covariance_
prec_ = model.precision_
lw_cov_, _ = ledoit_wolf(X)
lw_prec_ = linalg.inv(lw_cov_)
# #############################################################################
# Plot the results
plt.figure(figsize=(10, 6))
plt.subplots_adjust(left=0.02, right=0.98)
# plot the covariances
covs = [('Empirical', emp_cov), ('Ledoit-Wolf', lw_cov_),
('GraphicalLassoCV', cov_), ('True', cov)]
vmax = cov_.max()
for i, (name, this_cov) in enumerate(covs):
plt.subplot(2, 4, i + 1)
plt.imshow(this_cov, interpolation='nearest', vmin=-vmax, vmax=vmax,
cmap=plt.cm.RdBu_r)
plt.xticks(())
plt.yticks(())
plt.title('%s covariance' % name)
# plot the precisions
precs = [('Empirical', linalg.inv(emp_cov)), ('Ledoit-Wolf', lw_prec_),
('GraphicalLasso', prec_), ('True', prec)]
vmax = .9 * prec_.max()
for i, (name, this_prec) in enumerate(precs):
ax = plt.subplot(2, 4, i + 5)
plt.imshow(np.ma.masked_equal(this_prec, 0),
interpolation='nearest', vmin=-vmax, vmax=vmax,
cmap=plt.cm.RdBu_r)
plt.xticks(())
plt.yticks(())
plt.title('%s precision' % name)
if hasattr(ax, 'set_facecolor'):
ax.set_facecolor('.7')
else:
ax.set_axis_bgcolor('.7')
# plot the model selection metric
plt.figure(figsize=(4, 3))
plt.axes([.2, .15, .75, .7])
plt.plot(model.cv_alphas_, np.mean(model.grid_scores_, axis=1), 'o-')
plt.axvline(model.alpha_, color='.5')
plt.title('Model selection')
plt.ylabel('Cross-validation score')
plt.xlabel('alpha')
plt.show()
脚本的总运行时间:(0分0.755秒)




